Se encuentra usted aquí
Dr. Francisco Cubillos

Através de evolución experimental, el laboratorio del Dr Cubillos tiene por objetivo mejorar genéticamente levaduras previamente aisladas en Chile para fermentación. Esto es posible a través de la identificación de los cambios moleculares en levaduras responsable de la adaptación fisiológica a mostos de fermentación y que confluyen con procesos de domesticación en hongos. Para esto, se utilizan diversas técnicas Omicas y aproximaciones genómicas y bioinformáticas.
El Dr. Francisco A. Cubillos es Ingeniero en Biotecnología Molecular de la Universidad de Chile, desde donde egreso el año 2006. Posteriormente, realizó un Master en Ciencias Biológicas en la misma Institución, desde donde egreso el año 2008. El Dr. Cubillos completó sus estudios en la Universidad de Nottingham donde obtuvo su grado de Doctor el año 2010, siendo reconocido como el mejor estudiante de su generación.
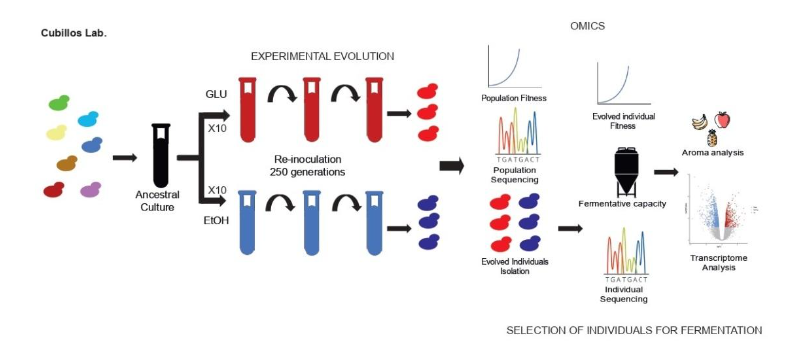
El Dr. Cubillos lidera actualmente el Laboratorio de Genética Molecular de la Universidad de Santiago de Chile como Investigador Independiente desde diciembre de 2015. Desde entonces, ha recibido cinco proyectos como investigador principal y ha publicado 13 manuscritos como autor correspondiente y 9 en colaboración con otros colegas en el campo. Hasta el momento, se graduaron diez estudiantes de pregrado y dos estudiantes de MRes. Su grupo de investigación actual incluye siete estudiantes de pregrado, cinco Ph.D. estudiantes y cuatro investigadores posdoctorales. Su investigación se centra en estudios filogenómicos en levaduras patagónicas tolerantes al etanol, con impactos científicos y biotecnológicos fundamentales. Además, su grupo se ha especializado en el estudio de los cambios moleculares en levaduras nativas derivados de procesos de evolución experimental con el objetivo de obtener levaduras mejoradas genéticamente para fermentación de cerveza, hidromiel, entre otros fermentados (ver figura). Su grupo está comprometido con la excelencia en la investigación en Chile y con el apoyo al desarrollo de nuevas aplicaciones biotecnológicas para productos fermentativos.
-
Enero 2021 - Diciembre 2024. EMBO Global Investigator. Phylogenomic studies in ethanol-tolerant yeast from Patagonia. Principal Investigator
-
Noviembre 2020 - Noviembre 2022. FIC Región de los Rios-Chile. Supporting economic re-activation of the brewing Industry in the Rios Region. Co-researcher
-
Enero 2020 – Enero 2022: Networking Grant CONICYT-PCI REDES for International Scientific Cooperation between iBio – Wisconsin Energy Institute (US) and Delft University Technology (Netherlands). ‘The SynVAR Collaborative Network: Yeast genetics for Synthetic Biology and Natural Variation advances in native Patagonian yeasts’. Principal Investigator.
-
Enero 2019 – Enero 2022: Research Grant for International Scientific Cooperation ECOS/CONICYT C18B03 ‘Genomic and physiological Characterization of ethanol-producing yeast populations from Patagonia’ between Universidad de Santiago de Chile and Sorbonne Université. Principal Researcher.
-
Mayo 2018 - Mayo 2020. FIC Región de los Rios. ‘Hacia la denominación de origen de la cerveza de Los Ríos: descubriendo una levadura nativa’. Co-Researcher
-
Abril 2018 – Marzo 2022. FONDECYT Regular 1180161 ‘Decoding the genomic and phenomic complexity of the cryotolerant yeast Saccharomyces eubayanus’. Principal Investigator.
-
Diciembre 2017 – Diciembre 2027: Millennium Institute for Integrative Biology (iBio). Associate Researcher.
-
Enero 2017 – Enero 2020: Grant INACH Regular RT_09_16 (2017-2020). ‘Biochemical and molecular responses disclose mechanisms of Antarctic macroalgae to thrive under Climate Change’. Co-Investigator.
-
Mayo 2016 - Noviembre 2017: Networking Grant CONICYT-PCI REDES for International Scientific Cooperation Universidad de Santiago de Chile – INRA Montpellier, France with Prof Sylvie Dequin. ‘Applied yeast genetics for high quality food products’. Principal Investigator.
-
Enero 2016 - Diciembre 2017: Research Grant Millennium Nucleus in Synthetic Fungi Biology MNFISB. Associate Researcher
1. Carlos A. Villarroel, Paulo Canessa, Macarena Bastias, Francisco A CubillosUncovering divergence in gene expression regulation in the adaptation of yeast to nitrogen scarcity. bioRxiv. 2021
2. Eeshit Dhaval Vaishnav, Carl G de Boer, Moran Yassour, Jennifer Molinet, Lin Fan, Xian Adiconis, Dawn A Thompson, Francisco A Cubillos, Joshua Z Levin, Aviv Regev. A comprehensive fitness landscape model reveals the evolutionary history and future evolvability of eukaryotic cis-regulatory DNA sequences. bioRxiv. 2021. doi: https://doi.org/10.1101/2021.02.17.430503
3. Wladimir Mardones, Carlos A. Villarroel, Valentina Abarca, Kamila Urbina, Tomás A. Peña, Jennifer Molinet, Roberto F. Nespolo and Francisco A. Cubillos*. Rapid selection response to ethanol in Saccharomyces eubayanus emulates the domestication process under brewing conditions. Microb Biotechnol. 2020. Accepted. Doi: 10.1111/(ISSN)1751-7915.
4. Jennifer Molinet and Francisco A. Cubillos. Wild yeast for the future: Exploring the use of wild strains for wine and beer fermentation. Front Genet. 2020 Nov 2;11:589350. doi: 10.3389/fgene.2020.589350. eCollection 2020.
5. Mandakovic D, Pulgar R, Maldonado J, Mardones W, González M, Cubillos FA*, Cambiazo V*. Fungal Diversity Analysis of Grape Musts from Central Valley-Chile and Characterization of Potential New Starter Cultures. Microorganisms. 2020 Jun 24;8(6):956. doi: 10.3390/microorganisms8060956
6. Urbina K*, Villarreal P*, Nespolo RF, Salazar R, Santander R, Cubillos FA. Volatile compound screening using HS-SPME-GC/MS on Saccharomyces eubayanus strains under low-temperature pilsner wort fermentation. Microorganisms 2020, 8(5), 755; https://doi.org/10.3390/microorganisms8050755.
7. Nespolo RF*, Villarroel CA*, Oporto CI, Tapia SM, Vega F, Urbina K, De Chiara M, Mozzachiodi S, Mikhalev E, Thompson D, Saenz-Agudelo P, Larrondo LF, Liti G, Cubillos FA. An Out-of-Patagonia migration explains the worldwide diversity and distribution of Saccharomyces eubayanus lineages. PLoS Genetics. 2020. 10.1371/journal.pgen.1008777.
8. Devia J., Bastias C., Kessi-Perez EI., Villarroedl CA, Cubillos FA, Liti G., Martínez C & Salinas F. Transcriptional activity and protein levels for horizontally acquired genes in yeast reveal hallmarks of adaptation to fermentative environments. Front Genet. 2020 Apr 30;11:293. doi: 10.3389/fgene.2020.00293. eCollection 2020.
9. Roberto F. Nespolo, Jaiber J. Solano-Iguaran, Rocío Paleo-López, Julian F. Quintero-Galvis, Francisco A. Cubillos, Francisco Bozinovic. Performance, genomic rearrangements and signatures of adaptive evolution: lessons from fermentative yeasts. Ecology and Evolution. 2020. Doi: 10.1002/ece3.6208.
10. Mardones W, Villarroel CA, Krogerus K, Tapia SM, Urbina K, Oporto CI, O'Donnell S, Minebois R, Nespolo R, Fischer G, Querol A, Gibson B, Cubillos FA*. Molecular profiling of beer wort fermentation diversity across natural Saccharomyces eubayanus isolates. Microb Biotechnol. 2020 Feb 25. doi: 10.1111/1751-7915.13545
11. Libkind D, Peris D, Cubillos FA, Steenwyk JL, Opulente DA, Langdon QK, Rokas A, Hittinger CT. Into the wild: new yeast genomes from natural environments and new tools for their analysis. FEMS Yeast Res. 2020 Feb 3. pii: foaa008. doi: 10.1093/femsyr/foaa008
12. Jennifer Molinet, Francisco A. Cubillos, Francisco Salinas, Gianni Liti and Claudio Martínez. Genetic variants of TORC1 signaling pathway affect nitrogen consumption in Saccharomyces cerevisiae during alcoholic fermentation. PLoS One 2019 Jul 26;14(7):e0220515. doi: 0.1371/journal.pone.0220515. eCollection 2019
13. Christian I. Oporto, Carlos A. Villarroel, Sebastián M. Tapia, Verónica García and Francisco A. Cubillos*. Distinct Transcriptional Changes in Response to Patulin Underlie Toxin Biosorption Differences in Saccharomyces cerevisiae. Toxins 2019, 11(7), 400; https://doi.org/10.3390/toxins11070400
14. Cubillos, FA., Gibson, B.*, Grijalva Vallejos, N., Krogerus, K., Nikulin, J. 2019. Bioprospecting for brewers: exploiting natural diversity for naturally diverse beers. Yeast. 2019 Jun;36(6):383-398. doi: 10.1002/yea.3380
15. Tapia SM, Cuevas M, Abarca V, Delgado V, Rojas V, García V, Brice C, Martínez C, Salinas F, Larrondo LF, Cubillos FA*. 2018. GPD1 and ADH3 natural variants underlie glycerol yield differences in wine fermentation. Frontiers in Microbiology. 9:1460. doi: 10.3389/fmicb.2018.01460.
16. Brice C, Cubillos FA, Dequin S, Camarasa C, Martínez C 2018. Adaptability of the Saccharomyces cerevisiae yeasts to wine fermentation conditions relies on their strong ability to consume nitrogen. PLoS One 2018 Feb 12;13(2):e0192383. doi: 10.1371/journal.pone.0192383. eCollection 2018.
17. Quispe X, Tapia SM, Villarroel C, Oporto C, Abarca V, García V, Martínez C, Cubillos FA*. 2017. Genetic basis of mycotoxin susceptibility differences between budding yeast isolates. Scientific Reports. 2017 Aug 23;7(1):9173. doi: 10.1038/s41598-017-09471-z.
18. Godoy L, Silva-Moreno E, Mardones W, Guzman D, Cubillos FA, Ganga A. 2017. Genomics Perspectives on Metabolism, Survival Strategies, and Biotechnological Applications of Brettanomyces bruxellensis LAMAP2480. J Mol Microbiol Biotechnol. 2017 Jun 9;27(3):147-158.
19. Cubillos FA, Brice C, Molinet J, Tisné S, Abarca V, Tapia SM, Oporto C, García V, Liti G & Martínez C. 2017. Identification of nitrogen consumption genetic variants in yeast through QTL mapping and Bulk segregant RNA-seq analyses. G3 (Bethesda). 2017 Jun 7;7(6):1693-1705.
20. Thompson DA & Cubillos FA*. 2017. Natural gene expression variation studies in yeast. Yeast. Jan;34(1):3-17.
21. Cubillos F.A*. Exploiting budding yeast natural variation for industrial processes. 2016. Current Genetics. Nov;62(4):745-75.
22. Kessi-Pérez EI, Araos S, García V, Salinas F, Abarca V, Larrondo LF, Martínez C & Cubillos FA*. 2016. RIM15 antagonistic pleiotropy is responsible for differences in fermentation and stress response kinetics in budding yeast. FEMS Yeast Research. pii: fow021. Epub 2016 Mar 4.
23. Salinas F, de Boer CG, Abarca V, García V, Cuevas M, Araos S, Larrondo LF, Martínez C, Cubillos FA*. 2016. Natural variation in non-coding regions underlying phenotypic diversity in budding yeast. Scientific Reports, srep21849.